Notas de R
2024-09-23
Chapter 1 Introducción a R
1.1 Historia de R
R es un lenguaje de programación derivado de S.
¿Qué es S? Es un lenguaje de programación desarrollado por John Chambers (AT&T) en 1976, su función inicial era el análisis estadístico. Fue hasta 1998 que se lanzó la cuarta versión en “Programming with Data”, dicho libro documenta una versión muy similar a la que conocemos hoy en día, la cual incluye el análisis funcional estadístio. El lenguaje S tiene como raíz el análisis de datos, sus desarrolladores se enfocaron en contruir un lenguaje que resultara sencillo tanto para los ellos como para los usuarios, ¿Cómo? desarrollando un método de programación basado en líneas de comando.
Volviendo a R, se creó en 1991 por Ross Ihaka y Robert Gentleman. Fue en 1996 que se lanzó oficialmente: “R: A lenguaje for data anlysis and graphics”, siendo incialmente bastante similar al lenguaje S. Una gran ventaja de R fue su lanzamiento como software libre (la limitación clave de S fue su única disponibilidad como paquete comercial, S-PLUS), esto permitió que la fuente de código del sistema entero fuera accesible para cualquiera que decidiera emplearlo.
Al día de hoy R se puede emplear en casi cualquier plataforma de cómputo y sistema operativo, esto es gracias a su naturaleza open source, es decir, que cualquiera es libre de adaptar el software a la plataforma que desee.
Uno de los más grandes beneficios de R, no relacionado per se al lenguaje, es la comunidad de usuarios. Sus característicad de una comunidad muy activa, multidisciplinaria y de distintas partes del mundo ha permitido la construcción de una plataforma que tiene éxito en medida que las personas crean y apoyan el desarrollo de nuevas herramientas, paquetes, aplicaciones, así mismo como el apoyo a nuevos usuarios.
1.2 Ventajas de R
R es un entorno integrado para el manejo de datos, el cálculo, la generación de gráficos y análisis estadísticos. Las principales ventajas del uso de R son:
- Software libre.
- Facilidad para el manejo y almacenamiento de datos.
- Un conjunto de operadores para el cálculo de vectores y matrices.
- Una colección extensa e integrada de herramientas intermedias para el análisis estadístico de datos.
- Multitud de facilidades gráficas de altísima calidad.
- Un lenguaje de programación (muy) poderoso con muchas librerías especializadas disponibles. CRAN tiene aproximadamente 10k paqueterías disponibles, muchas más que el número de funciones de Excel.
- La mejor herramienta para trabajar con datos genómicos, proteómicos, redes, metabolómica, entre varias más.
- Casi todos podemos aprender por nuestra cuenta a usar excel (pero hay que pagar por la licencia, es software privativo…). Sin embargo, aunque es más difícil aprender por nuestra cuenta R, si lo hacemos nos da una ventaja comparativa sobre el resto de estudiantes de licenciaturas afines.
- R tiene la capacidad de relacionarse y trabajar de manera paralela con otros software (Microsoft Office, QGIS..). Algunas ventajas de R sobre, por ejemplo, la paquetería Office, son su capacidad de iteración, reproducibilidad, automatización, la generación de reportes dinámicos, múltiples formatos de salida (PDF, HTML, páginas de internet, artículos científicos, diapositivas), conexión directa con buscadores de internet.
1.3 Learning R Hub
En este sitio web se presentan varios recurso adicionales en línea para aprender R
1.4 Introducción a R para Estudiantes de Microbiología
R es un lenguaje de programación y un entorno de desarrollo estadístico ampliamente utilizado en la comunidad científica, incluida la microbiología. Este poderoso recurso ofrece una variedad de ventajas y funcionalidades que pueden beneficiar significativamente a los estudiantes de microbiología en su investigación y análisis de datos.
1.4.1 Ventajas de Aprender R para Estudiantes de Microbiología
1. Análisis Estadístico Avanzado
R proporciona una amplia gama de paquetes y herramientas estadísticas que permiten a los estudiantes de microbiología realizar análisis avanzados de datos, desde pruebas de hipótesis básicas hasta modelos de regresión y análisis multivariados.
2. Visualización de Datos
Con paquetes como ggplot2, los estudiantes pueden crear visualizaciones de datos interactivas y de alta calidad que facilitan la comprensión de patrones y tendencias en conjuntos de datos microbiológicos, como datos de secuenciación genómica o datos de ecología microbiana.
3. Reproducibilidad y Documentación
RMarkdown es una herramienta poderosa que permite a los estudiantes escribir documentos reproducibles que combinan código, resultados y narrativa en un solo lugar. Esto promueve la transparencia, la reproducibilidad y una mejor documentación de los análisis microbiológicos.
4. Acceso a una Comunidad Activa
R cuenta con una comunidad activa de usuarios y desarrolladores que comparten código, paquetes y recursos educativos. Los estudiantes pueden aprovechar este recurso para buscar ayuda, colaborar en proyectos y mantenerse al tanto de las últimas tendencias en análisis de datos microbiológicos.
5. Flexibilidad y Personalización
R es un lenguaje altamente flexible que permite a los estudiantes adaptar sus análisis a las necesidades específicas de sus proyectos microbiológicos. Desde la manipulación de datos hasta la creación de modelos personalizados, R ofrece la libertad y la capacidad de personalización necesarias para abordar una amplia variedad de preguntas de investigación en microbiología.
Aprender R puede ser extremadamente beneficioso para los estudiantes de microbiología al proporcionarles las herramientas y habilidades necesarias para realizar análisis de datos sofisticados, visualizar resultados de manera efectiva y promover la reproducibilidad en su investigación científica.
1.5 Comienza con R
1.5.1 Instalación
Para iniciar en R es necesario instalarlo. R está disponible para los sistemas Windows, Mac OS X y Linux. El lenguaje de programación R tiene integrado un ambiente de desarrollo (IDE, por sus siglas en inglés) llamado RStudio. Este IDE facilita la sintaxis y edición del código, así como la visualización de objetos.
Si requiere ver un tutorial de cómo instalar R y RStudio para Mac o Windows, puede apoyarse de los siguientes videos:
1.5.2 Paquetes o bibliotecas
Las funciones especializadas de R se guardan en paquetes (packages) que deben ser invocados ANTES de llamar a una función del paquete.
Una manera de instalar paquetes es mediante el repositorio CRAN.
Navega por CRAN y encuentra algunos paquetes que podrían interesarte. Hay miles y cada día aumentan.
Para saber qué paquetes se tienen instalados en
tu máquina teclea la función library()
Para cargar un paquete, que se encuentre previamente instalado, se debe teclear
library(nombre_de_paquete)
Por ejemplo:
Para visualizar los paquetes ya cargados, teclea:
## [1] ".GlobalEnv" "package:ggpubr"
## [3] "package:palmerpenguins" "package:ggplot2"
## [5] "package:readxl" "package:gplots"
## [7] "tools:rstudio" "package:stats"
## [9] "package:graphics" "package:grDevices"
## [11] "package:utils" "package:datasets"
## [13] "package:methods" "Autoloads"
## [15] "package:base"Para visualizar las funciones dentro de un paquete en particular se utiliza:
## [1] "%>%" "add_summary"
## [3] "annotate_figure" "as_ggplot"
## [5] "as_npc" "as_npcx"
## [7] "as_npcy" "background_image"
## [9] "bgcolor" "border"
## [11] "change_palette" "clean_table_theme"
## [13] "clean_theme" "colnames_style"
## [15] "color_palette" "compare_means"
## [17] "create_aes" "desc_statby"
## [19] "diff_express" "facet"
## [21] "fill_palette" "font"
## [23] "gene_citation" "gene_expression"
## [25] "geom_bracket" "geom_exec"
## [27] "geom_pwc" "geom_signif"
## [29] "get_breaks" "get_coord"
## [31] "get_legend" "get_palette"
## [33] "get_summary_stats" "ggadd"
## [35] "ggadjust_pvalue" "ggarrange"
## [37] "ggballoonplot" "ggbarplot"
## [39] "ggboxplot" "ggdensity"
## [41] "ggdonutchart" "ggdotchart"
## [43] "ggdotplot" "ggecdf"
## [45] "ggerrorplot" "ggexport"
## [47] "gghistogram" "ggline"
## [49] "ggmaplot" "ggpaired"
## [51] "ggpar" "ggparagraph"
## [53] "ggpie" "ggpubr_options"
## [55] "ggqqplot" "ggscatter"
## [57] "ggscatterhist" "ggstripchart"
## [59] "ggsummarystats" "ggsummarytable"
## [61] "ggtext" "ggtexttable"
## [63] "ggviolin" "gradient_color"
## [65] "gradient_fill" "grids"
## [67] "group_by" "labs_pubr"
## [69] "mean_ci" "mean_range"
## [71] "mean_sd" "mean_se_"
## [73] "median_hilow_" "median_iqr"
## [75] "median_mad" "median_q1q3"
## [77] "median_range" "mutate"
## [79] "npc_to_data_coord" "rotate"
## [81] "rotate_x_text" "rotate_y_text"
## [83] "rownames_style" "rremove"
## [85] "set_palette" "show_line_types"
## [87] "show_point_shapes" "stat_anova_test"
## [89] "stat_bracket" "stat_central_tendency"
## [91] "stat_chull" "stat_compare_means"
## [93] "stat_conf_ellipse" "stat_cor"
## [95] "stat_friedman_test" "stat_kruskal_test"
## [97] "stat_mean" "stat_overlay_normal_density"
## [99] "stat_pvalue_manual" "stat_pwc"
## [101] "stat_regline_equation" "stat_stars"
## [103] "stat_welch_anova_test" "tab_add_border"
## [105] "tab_add_footnote" "tab_add_hline"
## [107] "tab_add_title" "tab_add_vline"
## [109] "tab_cell_crossout" "tab_ncol"
## [111] "tab_nrow" "table_cell_bg"
## [113] "table_cell_font" "tbody_add_border"
## [115] "tbody_style" "text_grob"
## [117] "thead_add_border" "theme_classic2"
## [119] "theme_cleveland" "theme_pubclean"
## [121] "theme_pubr" "theme_transparent"
## [123] "ttheme" "xscale"
## [125] "yscale"EJEMPLOS DE VISUALIZACIÓN DE GRÁFICOS
##
##
## demo(graphics)
## ---- ~~~~~~~~
##
## > # Copyright (C) 1997-2009 The R Core Team
## >
## > require(datasets)
##
## > require(grDevices); require(graphics)
##
## > ## Here is some code which illustrates some of the differences between
## > ## R and S graphics capabilities. Note that colors are generally specified
## > ## by a character string name (taken from the X11 rgb.txt file) and that line
## > ## textures are given similarly. The parameter "bg" sets the background
## > ## parameter for the plot and there is also an "fg" parameter which sets
## > ## the foreground color.
## >
## >
## > x <- stats::rnorm(50)
##
## > opar <- par(bg = "white")
##
## > plot(x, ann = FALSE, type = "n")
##
## > abline(h = 0, col = gray(.90))
##
## > lines(x, col = "green4", lty = "dotted")
##
## > points(x, bg = "limegreen", pch = 21)
##
## > title(main = "Simple Use of Color In a Plot",
## + xlab = "Just a Whisper of a Label",
## + col.main = "blue", col.lab = gray(.8),
## + cex.main = 1.2, cex.lab = 1.0, font.main = 4, font.lab = 3)
##
## > ## A little color wheel. This code just plots equally spaced hues in
## > ## a pie chart. If you have a cheap SVGA monitor (like me) you will
## > ## probably find that numerically equispaced does not mean visually
## > ## equispaced. On my display at home, these colors tend to cluster at
## > ## the RGB primaries. On the other hand on the SGI Indy at work the
## > ## effect is near perfect.
## >
## > par(bg = "gray")
##
## > pie(rep(1,24), col = rainbow(24), radius = 0.9)
##
## > title(main = "A Sample Color Wheel", cex.main = 1.4, font.main = 3)
##
## > title(xlab = "(Use this as a test of monitor linearity)",
## + cex.lab = 0.8, font.lab = 3)
##
## > ## We have already confessed to having these. This is just showing off X11
## > ## color names (and the example (from the postscript manual) is pretty "cute".
## >
## > pie.sales <- c(0.12, 0.3, 0.26, 0.16, 0.04, 0.12)
##
## > names(pie.sales) <- c("Blueberry", "Cherry",
## + "Apple", "Boston Cream", "Other", "Vanilla Cream")
##
## > pie(pie.sales,
## + col = c("purple","violetred1","green3","cornsilk","cyan","white"))
##
## > title(main = "January Pie Sales", cex.main = 1.8, font.main = 1)
##
## > title(xlab = "(Don't try this at home kids)", cex.lab = 0.8, font.lab = 3)
##
## > ## Boxplots: I couldn't resist the capability for filling the "box".
## > ## The use of color seems like a useful addition, it focuses attention
## > ## on the central bulk of the data.
## >
## > par(bg="cornsilk")
##
## > n <- 10
##
## > g <- gl(n, 100, n*100)
##
## > x <- rnorm(n*100) + sqrt(as.numeric(g))
##
## > boxplot(split(x,g), col="lavender", notch=TRUE)
##
## > title(main="Notched Boxplots", xlab="Group", font.main=4, font.lab=1)
##
## > ## An example showing how to fill between curves.
## >
## > par(bg="white")
##
## > n <- 100
##
## > x <- c(0,cumsum(rnorm(n)))
##
## > y <- c(0,cumsum(rnorm(n)))
##
## > xx <- c(0:n, n:0)
##
## > yy <- c(x, rev(y))
##
## > plot(xx, yy, type="n", xlab="Time", ylab="Distance")
##
## > polygon(xx, yy, col="gray")
##
## > title("Distance Between Brownian Motions")
##
## > ## Colored plot margins, axis labels and titles. You do need to be
## > ## careful with these kinds of effects. It's easy to go completely
## > ## over the top and you can end up with your lunch all over the keyboard.
## > ## On the other hand, my market research clients love it.
## >
## > x <- c(0.00, 0.40, 0.86, 0.85, 0.69, 0.48, 0.54, 1.09, 1.11, 1.73, 2.05, 2.02)
##
## > par(bg="lightgray")
##
## > plot(x, type="n", axes=FALSE, ann=FALSE)
##
## > usr <- par("usr")
##
## > rect(usr[1], usr[3], usr[2], usr[4], col="cornsilk", border="black")
##
## > lines(x, col="blue")
##
## > points(x, pch=21, bg="lightcyan", cex=1.25)
##
## > axis(2, col.axis="blue", las=1)
##
## > axis(1, at=1:12, lab=month.abb, col.axis="blue")
##
## > box()
##
## > title(main= "The Level of Interest in R", font.main=4, col.main="red")
##
## > title(xlab= "1996", col.lab="red")
##
## > ## A filled histogram, showing how to change the font used for the
## > ## main title without changing the other annotation.
## >
## > par(bg="cornsilk")
##
## > x <- rnorm(1000)
##
## > hist(x, xlim=range(-4, 4, x), col="lavender", main="")
##
## > title(main="1000 Normal Random Variates", font.main=3)
##
## > ## A scatterplot matrix
## > ## The good old Iris data (yet again)
## >
## > pairs(iris[1:4], main="Edgar Anderson's Iris Data", font.main=4, pch=19)
##
## > pairs(iris[1:4], main="Edgar Anderson's Iris Data", pch=21,
## + bg = c("red", "green3", "blue")[unclass(iris$Species)])
##
## > ## Contour plotting
## > ## This produces a topographic map of one of Auckland's many volcanic "peaks".
## >
## > x <- 10*1:nrow(volcano)
##
## > y <- 10*1:ncol(volcano)
##
## > lev <- pretty(range(volcano), 10)
##
## > par(bg = "lightcyan")
##
## > pin <- par("pin")
##
## > xdelta <- diff(range(x))
##
## > ydelta <- diff(range(y))
##
## > xscale <- pin[1]/xdelta
##
## > yscale <- pin[2]/ydelta
##
## > scale <- min(xscale, yscale)
##
## > xadd <- 0.5*(pin[1]/scale - xdelta)
##
## > yadd <- 0.5*(pin[2]/scale - ydelta)
##
## > plot(numeric(0), numeric(0),
## + xlim = range(x)+c(-1,1)*xadd, ylim = range(y)+c(-1,1)*yadd,
## + type = "n", ann = FALSE)
##
## > usr <- par("usr")
##
## > rect(usr[1], usr[3], usr[2], usr[4], col="green3")
##
## > contour(x, y, volcano, levels = lev, col="yellow", lty="solid", add=TRUE)
##
## > box()
##
## > title("A Topographic Map of Maunga Whau", font= 4)
##
## > title(xlab = "Meters North", ylab = "Meters West", font= 3)
##
## > mtext("10 Meter Contour Spacing", side=3, line=0.35, outer=FALSE,
## + at = mean(par("usr")[1:2]), cex=0.7, font=3)
##
## > ## Conditioning plots
## >
## > par(bg="cornsilk")
##
## > coplot(lat ~ long | depth, data = quakes, pch = 21, bg = "green3")
##
## > par(opar)Ejercicios
- Instala las siguientes librerías que te serviran durante todo el curso
- markdown
- ggplot2
INFORMACIÓN ADICIONAL
Existen repositorios adicionales a CRAN, uno de ellos es Bioconductor, en él puedes buscar e instalar paquetes como ggtree.
Otra plataforma que resulta de gran apoyo es GitHub, permite crear, almacenar, administrar y compartir códigos de distintos lenguajes de programación. Una de sus ventajas es la consulta de repositorios, por ejemplo mixOmics, el cual contiene una amplia variedad de métodos para la exploración e integración de datos biológicos. El paquete mixOmics contiene una gran cantidad de técnicas multivariadas que se han desarrollado y validado en múltiples estudios biológicos, esto mediante la implementación simultánea de distintas “ómicas” para obtener una mejor compresión del sistema.
Ejercicio
1. Explora la página de Bioconductor, apóyate de su buscador e instala el paquete ggtree.
1.6 Ayuda en R
En la mayoría de las ocasiones desconocemos el alcance de alguna paquetería, los criterios de alguna función o en general, sabemos lo que queremos hacer pero no tenemos ni idea de qué paqueteria usar.
Los comandos help() y ? son equivalentes, ambos van a permitir encontrar información sobre paqueterias, comandos o funciones generales de R. Se debe teclear help(nombre_comando) o ?nombre_comando
Por ejemplo, para buscar información detallada del comando solve:
Para buscar ayuda de funciones o palabra reservadas se utilizan comillas:
También existen opciones como help.start() y help.search() para obtener una versión extendida de la ayuda general desplegada en un navegador. Para ello se requiere tener la ayuda en HTML instalada y conexión a la red.
help.search() es una función que escanea documentación para paquetes previamente instalados.
Ejemplo:
help.start() es una función que despliega información basada en documentos en línea de la versión actual de R, además de brindar links a manuales y la lista de las paqueterías instaladas, entre otras cosas.
Ejemplo:
Cuando queremos ver ejemplos del uso de los comandos usamos la función example()
Ejemplo:
##
## hclust> require(graphics)
##
## hclust> ### Example 1: Violent crime rates by US state
## hclust>
## hclust> hc <- hclust(dist(USArrests), "ave")
##
## hclust> plot(hc)
##
## hclust> plot(hc, hang = -1)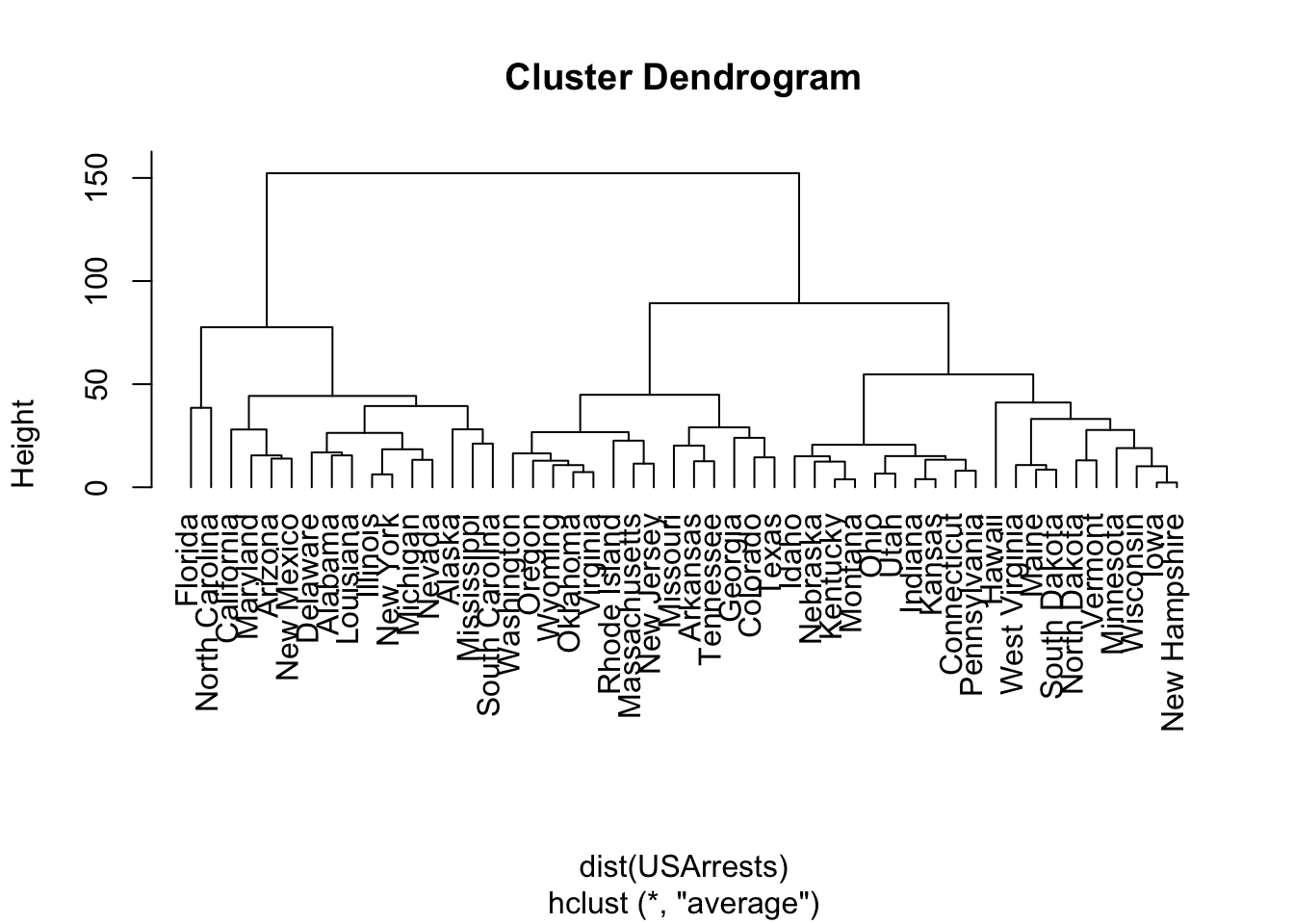
##
## hclust> ## Do the same with centroid clustering and *squared* Euclidean distance,
## hclust> ## cut the tree into ten clusters and reconstruct the upper part of the
## hclust> ## tree from the cluster centers.
## hclust> hc <- hclust(dist(USArrests)^2, "cen")
##
## hclust> memb <- cutree(hc, k = 10)
##
## hclust> cent <- NULL
##
## hclust> for(k in 1:10){
## hclust+ cent <- rbind(cent, colMeans(USArrests[memb == k, , drop = FALSE]))
## hclust+ }
##
## hclust> hc1 <- hclust(dist(cent)^2, method = "cen", members = table(memb))
##
## hclust> opar <- par(mfrow = c(1, 2))
##
## hclust> plot(hc, labels = FALSE, hang = -1, main = "Original Tree")##
## hclust> plot(hc1, labels = FALSE, hang = -1, main = "Re-start from 10 clusters")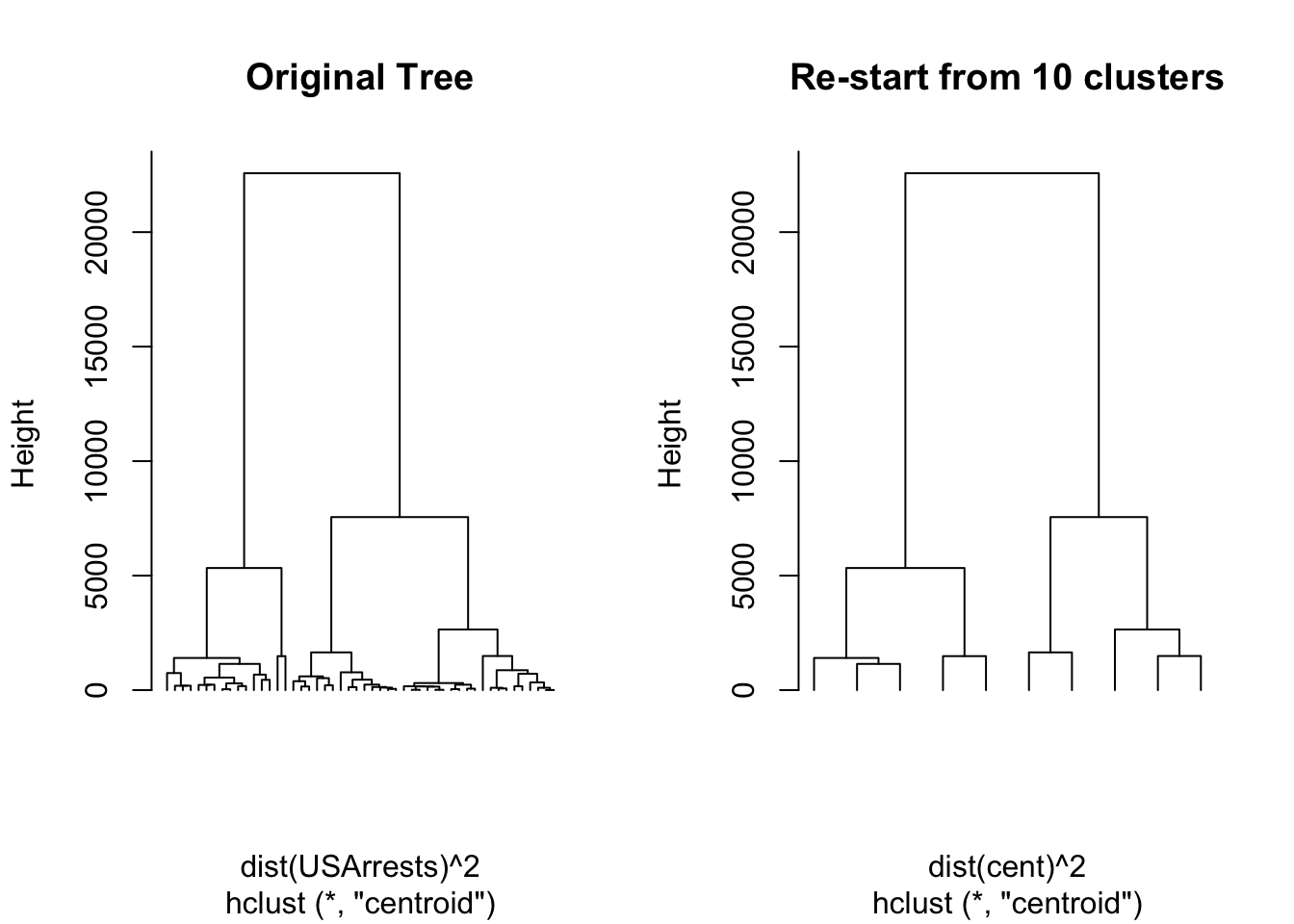
##
## hclust> par(opar)
##
## hclust> ### Example 2: Straight-line distances among 10 US cities
## hclust> ## Compare the results of algorithms "ward.D" and "ward.D2"
## hclust>
## hclust> mds2 <- -cmdscale(UScitiesD)
##
## hclust> plot(mds2, type="n", axes=FALSE, ann=FALSE)
##
## hclust> text(mds2, labels=rownames(mds2), xpd = NA)
##
## hclust> hcity.D <- hclust(UScitiesD, "ward.D") # "wrong"
##
## hclust> hcity.D2 <- hclust(UScitiesD, "ward.D2")
##
## hclust> opar <- par(mfrow = c(1, 2))
##
## hclust> plot(hcity.D, hang=-1)##
## hclust> plot(hcity.D2, hang=-1)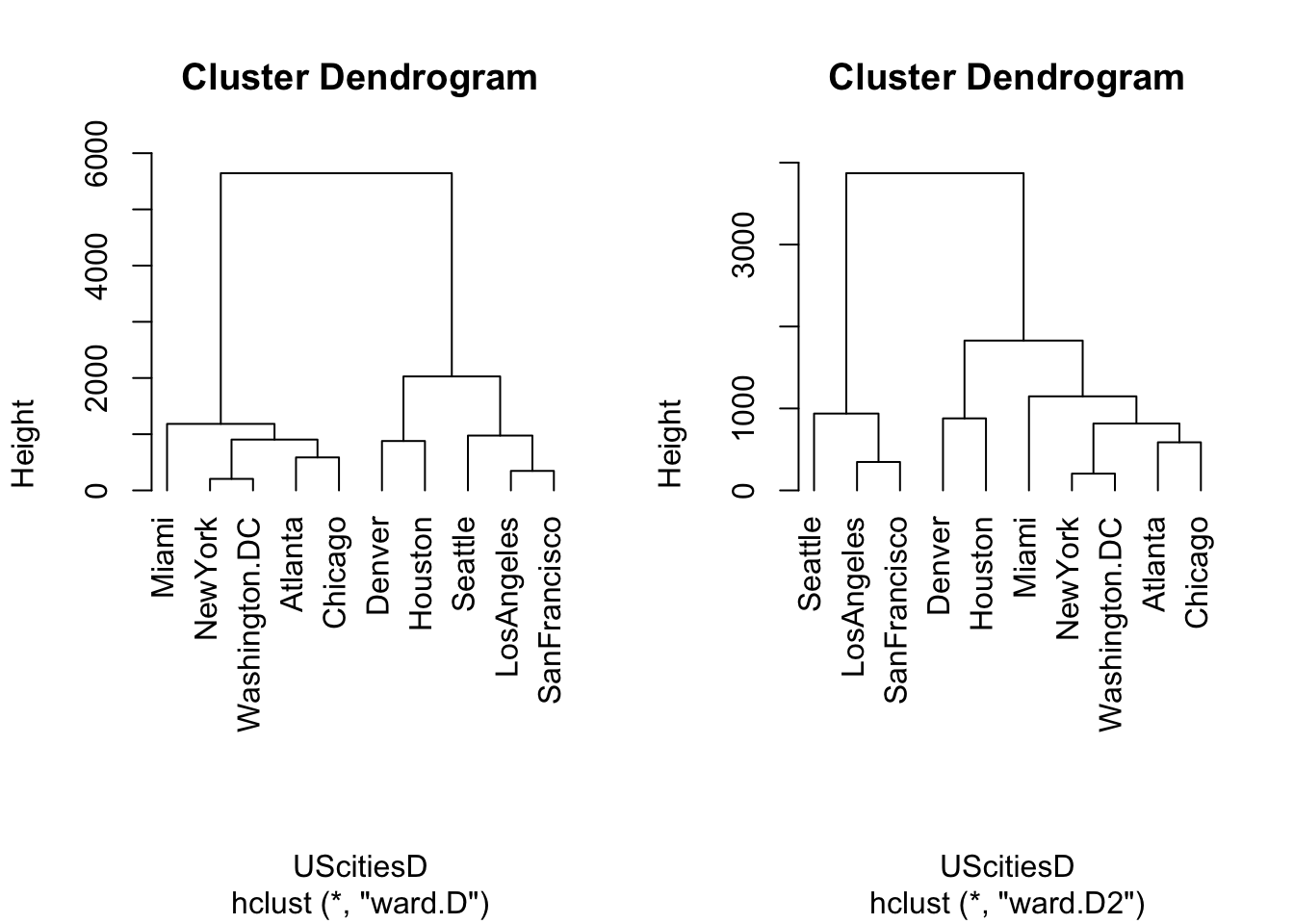
##
## hclust> par(opar)Todo lo anterior requiere que conozcamos el nombre correcto del comando, pero ¿qué pasa si no lo sabemos?, ¿lloramos? no.
Podemos utilizar el comando apropos() para encontrar todo lo relacionado con algún término.
Ejemplo:
## [1] ".rs.markdown.resolveCompletionRoot" ".rs.resolveAliasedPath"
## [3] ".rs.resolveAliasedSymbol" ".rs.resolveContextSourceRefs"
## [5] ".rs.resolveEnvironment" ".rs.resolveFormals"
## [7] ".rs.resolveFormalsImpl" ".rs.resolveFormalsImplS3Dispatch"
## [9] ".rs.resolveObjectFromFunctionCall" ".rs.resolveObjectSource"
## [11] ".rs.reticulate.resolveModule" ".rs.rnb.resolveActiveChunkId"
## [13] "backsolve" "forwardsolve"
## [15] "qr.solve" "solve"
## [17] "solve.default" "solve.qr"Ahora, ¿qué pasa cuando tengo la idea de lo que quiero hacer pero no se qué paqueteria usar, ni cuál comando? puedo usar ?? seguido de una palabra clave. Esto nos arrojará sugerencias sobre lo que deseamos hacer.
Ejemplo:
NOTA Se recomienda el uso del autocompletado, de esta manera reducirás errores de dedo.